| zurück | Histon | |||
| allgemeines | Basische Proteine des Zellkerns. | |||
| Funktion | Stabilisiert die DNA durch Bildung von Nukleosomen. Da die DNA eine Säureüberschuß hat, verbindet sie sich gut mit den basischen Histonen. | |||
| Nukleosomen | Ein Nukleosom besteht aus 8 Histonmolekülen, um die ein DNA - Abschnitt gewickelt ist. | |||
| Haupt-Histon-Proteine | H1, H2A, H2B, H3, H4 |
H2A und H2B bilden Dimere. H3 und H4 bilden ebenfalls Dimere. Zwei H3/H4-Dimere lagern sich zu einem Tetramer zusammen, an das wiederum zwei H2A/H2B-Dimere angelagert werden. Dadurch entsteht der oktamere Nukleosomenkern (´core particle´), um den sich die DNA in ca. zwei großen linksgängigen Windungen legen kann. Das fünfte Histon, H1, wird möglicherweise benötigt, um eine 30 nm-Faser zu bilden - eine übergeordnete Struktur, die einer Helix aus Nukleosomen entspricht. Dadurch wird die DNA Packung weiter verstärkt (1). 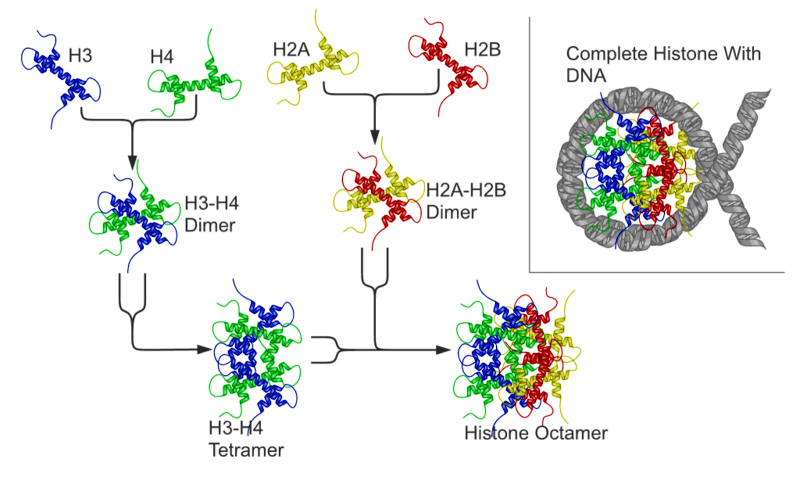 Bild aus Wikipedia 2012 (1) |
||
| H1 | Im Gegensatz zu H2A, H2B, H3 und H4 ist das Histon H1 in seiner Struktur sehr variabel. Bei einigen Arten fehlt es. | |||
| Makro-H2A | Ersetzt das Histon H2A partiell auf dem inaktivierten X-Chromosom von weiblichen Säugern. | |||
| CENP-A | Variante des Histons H3. Nur im Bereich des Zentromers zu finden. | |||
| Nukleosomen - Remodelling | Die DNA ist durch die Bindung an Histone blockiert. | Für Transskription, Repikation und Reparatur wird das Histon von der DNA abgelöst oder auf dem DNA-Strang verschoben. | ||
| Histon - Modifikationen | Methylierung, Phosphorylierung, Sumoylierung, Ubiquitinylierung, Acetylierung (HDAC). Teil der epigenetischen Genregulierung. | |||
| Acetylierung | Histon - Enden sind normalerweise durch Amin-Gruppen von Lysin und Arginin positiv geladen. | Dadurch kann Histon mit den negativ geladenen Phosphat - Gruppen der DNA reagieren. | Durch Acetylierung werden die Amin-Gruppen neutralisiert. | Die geringer gebundene DNA kann expandieren und Gene transscibieren. |
| Deacetylierung | HDAC3, HDAC11 | Durch entfernen von Acetyl - Gruppen wird DNA fester an Histon gebunden und die Transscription gebremst. | ||
| LSD1 | Lysine-spezifische Demethylase - 1, auch KDM1A. Inhibitor: Tranylcypromin (TCP, Antdepressivum). TCP kann eine Therapie der AML mit ATRA wirksam machen(2). Ohne TCP wirkt ATRA nur bei der APL. | |||
| Histon-mRNA | Hat an ihrem Ende eine haarnadelförmige Struktur befindet, welche die RNA stabilisiert. | Histone-mRNA wird am Ende der S-Phase oder bei pharmakologischer Hemmung der Replikation abgebaut. | ||
| Eri1 | Am Abbau von Histon-mRNA beteiligt(3). | exoRNase | inhibiert RNA - Interference | trimmt das 5.8S rRNA 3' -Ende. |
| Quellen |
1.) Histon. Wikipedia 2012 2.) Schenk T, et al.: Inhibition of the LSD1 (KDM1A) demethylase reactivates the all-trans-retinoic acid differentiation pathway in acute myeloid leukemia. Nature Medicine 18(2012):605–611 3.) Hoefig, K. et al.: Eri1 degrades the stem-loop of oligouridylated histone mRNAs to induce replication-dependent decay. Nat Struct Mol Biol 20(2013):73-81. doi: 10.1038/nsmb.2450, 2013 | |||
Impressum .....................................................................................Zuletzt geändert am 17.02.2013 21:52